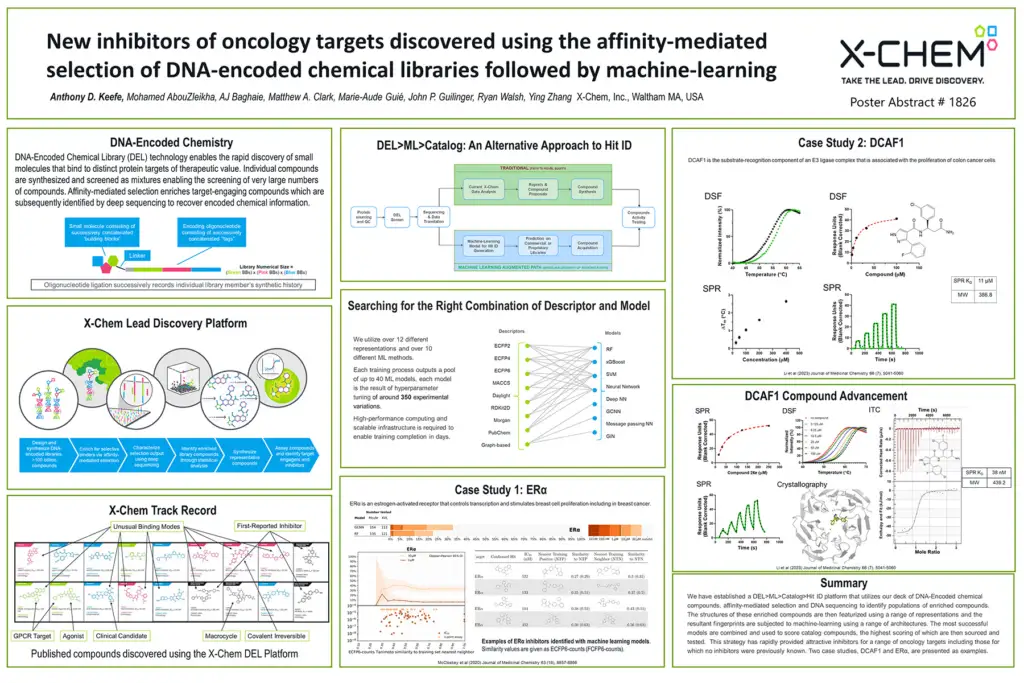
La plateforme DNA-Encoded Chemical Library (DEL) de X-Chem génère une grande quantité de données de liaison pour chaque cible contre laquelle elle est criblée. Nous présentons ici l'application de l'apprentissage automatique à ces grands ensembles de données avec des études de cas qui comprennent l'identification de nouveaux inhibiteurs de deux cibles oncologiques. La première, ERα, est un récepteur activé par les œstrogènes qui contrôle la transcription et stimule la prolifération des cellules mammaires, y compris dans le cancer du sein. Le second, DCAF1, est le composant de reconnaissance de substrat d'un complexe E3 ligase associé à la prolifération des cellules cancéreuses du côlon. Plus de 100 milliards de composés codés dans l'ADN ont été synthétisés et analysés pour déterminer leur affinité avec chaque cible. Les données de sortie de la sélection ont ensuite été regroupées et utilisées pour former plusieurs modèles d'apprentissage automatique. Les modèles dont le pouvoir prédictif a été démontré ont ensuite été utilisés pour classer les composés du catalogue virtuel qui ont ensuite été acquis et testés. Une série de stratégies de science des données réussies sera décrite, y compris celles qui ont conduit à des inhibiteurs à deux chiffres de nM de DCAF1 et ERα.
Poster présenté à la conférence de l'Association américaine pour la recherche sur le cancer (2024)