Le criblage de chimiothèques codées dans l'ADN est un excellent outil pour identifier de nouveaux liants adaptés aux dégradateurs bispécifiques
La valeur de la DEL pour les PROTACs et autres bispécifiques
Les chimères ciblant la protéolyse (PROTAC) sont des molécules bispécifiques qui induisent la dégradation protéique d'une cible spécifique. Depuis leur découverte il y a une vingtaine d'années, ces composés ont suscité un vif intérêt dans les domaines de la chimie et de la pharmacie.
En raison de leur structure, les PROTAC ont longtemps été considérés comme l'application parfaite pour le criblage de bibliothèques codées par l'ADN (DEL). Les PROTACS ont des structures modulaires, composées d'une partie liant la cible, d'un linker et d'un engageur avec une ligase E3. Ce qui rend la DEL parfaite pour créer cette structure bispécifique, c'est que dans la bibliothèque, le point de liaison est déjà intégré, reliant le composé découvert à son étiquette d'ADN. En fait, vous savez même que le linker est toléré dans l'événement de liaison parce qu'il était là lorsque vous avez découvert l'interaction. Il vous suffit donc de remplacer l'ADN par une ligase E3 appropriée pour que votre découverte soit prête.
Découverte de dégradateurs bispécifiques du récepteur α des œstrogènes avec de nouveaux liants
Pour démontrer ce concept, notre équipe a construit un dégradeur bispécifique (PROTAC) du récepteur des œstrogènes. α (ERα) dans le but d'améliorer les modulateurs actuels de l'ERα (inhibiteurs de l'aromatase/SERMs/SERDs) qui sont utilisés pour traiter le cancer du sein ER+. (Pour plus de détails, voir notre article, Dégradeurs bispécifiques du récepteur α des œstrogènes incorporant de nouveaux liants identifiés à l'aide d'un criblage de chimiothèques codées sur l'ADN juste à l'extérieur de la Journal of Medicinal Chemistry.)
Nous avons passé au crible environ 120 milliards de molécules codées dans l'ADN par rapport à l'ER de type sauvage.α et trois mutants à perte de fonction (GOF) conférant une résistance partielle aux thérapies endocriniennes existantes (Fig. 1a). Une fois que les petites molécules enrichies ont été identifiées grâce à leurs séquences d'ADN, les petites molécules liantes ont été rapidement exploitées dans un réseau parallèle de chimie click produisant des PROTACs pour ERα avec une variété de liants E3 (Fig. 1b).
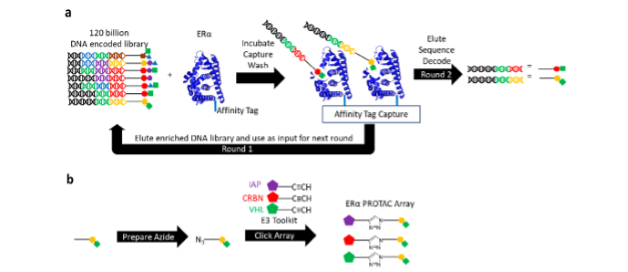
Figure 1. Représentation schématique des plateformes de criblage de chimiothèques encodées dans l'ADN (DECL) et de réseaux PROTAC. (a) Une DECL (petites molécules synthétisées de manière combinatoire et attachées à des étiquettes d'ADN) est incubée avec ERα (protéine cible, sélectionnée par affinité et lavée pour éliminer les non-liants). La liaison et la capture d'affinité sont répétées en utilisant l'éluat du premier tour comme intrant pour enrichir davantage la bibliothèque. Les étiquettes d'ADN de la sélection du cycle 2 sont séquencées pour identifier les petites molécules qui se lient. (b) Des petites molécules liantes liées à des variantes d'azide sont préparées et réagissent dans une bibliothèque parallèle de chimie click d'alcynes liants E3 afin d'obtenir un ensemble de PROTAC pour ERα.
Reproduit avec l'autorisation de Disch J.S. et al, Journal de la chimie médicinale 2021 64 (8), 5049-5066, DOI : 10.1021/acs.jmedchem.1c00127. Copyright © (2021) American Chemical Society.
Le criblage simultané permet d'obtenir deux composés puissants hors ADN, enrichis contre le type sauvage et les trois mutants GOF.
Comme la sélection de la bibliothèque DEL ne fournit pas seulement des résultats, mais des profils de résultats pour plusieurs cibles incluses dans l'expérience, elle permet de gagner beaucoup de temps. Dans ce cas, l'écran de découverte primaire a permis d'identifier deux composés puissants qui ont été enrichis contre l'ER de type sauvage.α et trois des mutants les plus courants - S463P, Y537S et D538G (Fig. 2a & b). En outre, les deux composés n'étaient pas enrichis en présence d'estradiol, ce qui indique qu'ils se lient probablement à la poche de liaison de l'estradiol. Pour confirmer les résultats de la sélection, des analogues directs hors ADN des deux composés puissants ont été synthétisés et leur affinité de liaison a été évaluée dans un essai de liaison par polarisation de fluorescence (Fig. 2c).
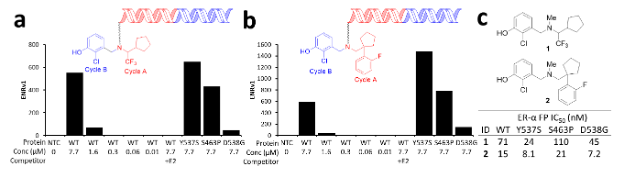
Figure 2. Confirmation de l'activité de la sortie DECL et des analogues hors ADN de deux membres apparentés. (a, b) Les profils d'enrichissement (ENRv1) de deux membres structurellement apparentés issus d'une bibliothèque à deux cycles montrent qu'ils se lient à l'ERα WT et aux mutants Y537S, S463P et D538G, mais ne se lient pas à la matrice (NTC ; pas de contrôle de la cible). Leur liaison à la WT est spécifiquement concurrencée par l'ajout du concurrent Estradiol (E2). Cela suggère que les membres de la bibliothèque interagissent au niveau du site de liaison du ligand. (c) Des exemples analogues synthétisés “sans ADN” (avec un linker et un ADN tronqué à un seul groupe méthyle) se lient dans un essai de déplacement de liaison par polarisation de fluorescence. Les résultats de l'essai sont une moyenne des essais en double.
Reproduit avec l'autorisation de Disch J.S. et al, Journal de la chimie médicinale 2021 64 (8), 5049-5066, DOI : 10.1021/acs.jmedchem.1c00127. Copyright © (2021) American Chemical Society.
S'assurer que les nouveaux composés se lient de manière antagoniste et non agoniste
Même si l'objectif de cette thérapie bispécifique est de se lier et de provoquer la dégradation, il est important de se rappeler que le liant à petite molécule peut avoir un effet cellulaire par lui-même. Dans ce cas, les liants que nous avons découverts se sont révélés être des agonistes lorsqu'ils ont été dosés dans les cellules. Nous avons pu convertir nos liants en antagonistes, en apportant de petites modifications à la structure pour établir une relation structure-activité (SAR) qui distingue l'antagonisme de l'agonisme.
Grâce à la chimie click sur nos antagonistes les plus actifs, nous avons produit une série de composés bispécifiques avec des éléments de liaison E3 variables et des longueurs de liens variables. Les tests de dégradation d'ERα dans des cellules humaines de cancer du sein (MCF7) ont révélé deux candidats PROTAC prometteurs : 18 et 21. Ces deux composés engagent VHL en tant que composant E3 ; les composés engageant IAP et CRBN n'ont pas démontré une dégradation comparable. Les deux 18 et 21 a démontré une liaison puissante de l'ERα et les trois mutants GOF (Fig. 3), dégradent ERα, entraînent des défauts de viabilité de MCF7 et régulent à la baisse l'expression de PGR.
Malgré la faible perméabilité, la solubilité limitée et la forte liaison aux protéines plasmatiques du 18 et 21, Ces deux PROTACs ont atteint des expositions plasmatiques significatives lorsqu'ils sont administrés par voie sous-cutanée (Fig. 3f), ce qui a justifié l'évaluation de leurs propriétés ERα de dégradation in vivo.
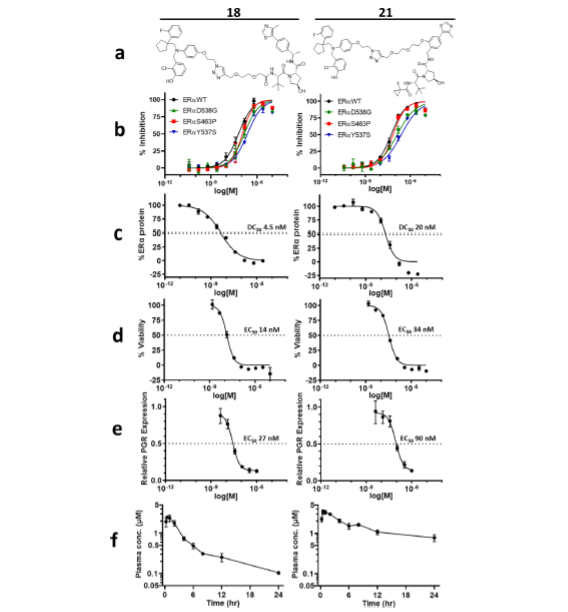
Figure 3. Propriétés biochimiques, cellulaires et pharmacocinétiques chez la souris des ERα PROTACs, 18 et 21. (a) Structures de 18 et 21. (b) Inhibition biochimique à l'aide d'ERα WT recombinant purifié et des mutants D538G, Y537Y et S463P. (c) Dégradation d'ERα dans les cellules MCF7 traitées pendant 24 h, suivie d'une analyse Western intracellulaire de la protéine ERα normalisée par rapport à la tubuline. Les graphiques représentent les niveaux ERα normalisés. (d) Essai de viabilité MCF7 avec des cellules traitées pendant 7 jours suivi d'un point final de viabilité par CellTiter-Glo. (e) Analyse de l'expression du récepteur de la progestérone (PGR) en aval dans les cellules MCF7 traitées pendant 24 heures, suivie d'une extraction de l'ARN, d'une synthèse du premier brin et d'une analyse qPCR pour l'estimation de l'ARN du récepteur de la progestérone, normalisée par rapport à la TBP (protéine de liaison de Tata) ; les graphiques représentent les niveaux normalisés de PGR. (f) Concentration plasmatique moyenne (μM) pour un dosage sous-cutané de 10 mpk (mg/kg) chez la souris CD1 (n = 3). Les courbes indiquées proviennent d'une seule série avec des réplicats techniques.
Reproduit avec l'autorisation de Disch J.S. et al, Journal de la chimie médicinale 2021 64 (8), 5049-5066, DOI : 10.1021/acs.jmedchem.1c00127. Copyright © (2021) American Chemical Society.
Test d'élimination des tumeurs in vivo
Nous avons comparé l'efficacité des composés 18 et 21 avec celle du Fulvestrant, traitement standard, dans un modèle de xénogreffe MCF7 de type sauvage, œstrogéno-dépendante (Fig. 4). Composé 21 a diminué le volume de la tumeur presque aussi bien que le Fulvestrant. Ces résultats démontrent que le 21 est excellent pour l'inhibition et la dégradation de l'ERα, à la fois in vitro et in vivo. La liaison ERα de 21 a été identifié directement à partir de l'écran DEL avec une optimisation minimale.
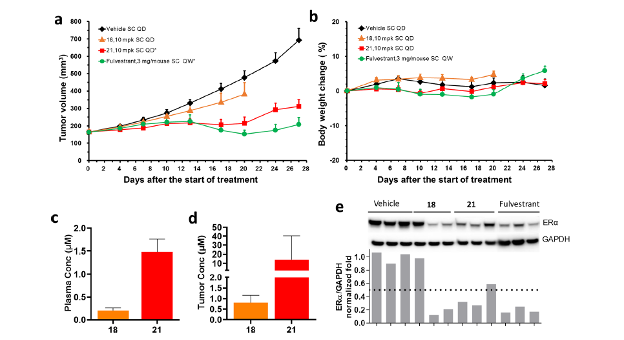
Figure 4. In vivo L'efficacité du produit est démontrée par une étude de l'efficacité du produit et une étude pharmacocinétique et pharmacodynamique (PK/PD) montrant l'efficacité du produit dans le modèle de xénogreffe MCF7. (a) Comparaison de l'activité antitumorale du 18 et 21 au Fulvestrant dans le modèle de xénogreffe MCF7 œstrogéno-dépendante WT. Composés 18 et 21 ont été administrés par voie sous-cutanée (SC) une fois par jour (QD) à raison de 10 mg/kg. Le fulvestrant a été administré par voie sous-cutanée une fois par semaine (QW) à raison de 3 mg/souris. Les données représentent le volume tumoral moyen ± SEM (n = 8). *p < 0,05 par rapport au contrôle du véhicule (test t de Student). (b) Tous les composés ont été bien tolérés sans modification du poids corporel. (c, d) La pharmacocinétique plasmatique terminale et la pharmacocinétique tumorale de 18 et 21 3 heures après la dernière dose (jour 20 ; 18) et (jour 27 ; 21). (e) Analyse PD de souris porteuses de tumeurs traitées avec les composés mentionnés ci-dessus pendant 3 jours. Une analyse par Western blot a été réalisée pour démontrer la réduction des niveaux d'ERα lors du traitement avec les composés mentionnés ci-dessus pendant 3 jours. 18, 21, et Fulvestra.
Reproduit avec l'autorisation de Disch J.S. et.al, Journal de la chimie médicinale 2021 64 (8), 5049-5066, DOI : 10.1021/acs.jmedchem.1c00127. Copyright © (2021) American Chemical Society.
X-Chem peut vous aider à identifier et à assembler les liants, les linkers et les ligands E3 pour votre prochain projet PROTAC
Ce cas démontre le potentiel et la puissance du DEL pour les applications PROTAC. En tant que leader de la technologie DEL, X-Chem peut vous aider à cribler des chimiothèques encodées dans l'ADN pour identifier de nouveaux binders, comme nous l'avons fait ici, et vous mettre sur la voie de la découverte d'un composé révolutionnaire. De l'identification de cibles difficiles au profilage biochimique et biophysique, en passant par l'élucidation du SAR grâce à la chimie médicinale et synthétique, nous sommes là pour vous aider dans votre quête des thérapies de demain qui sauveront des vies. Explorez notre site web pour en savoir plus.
Référence
- Disch, J.S. et al. Dégradeurs bispécifiques du récepteur α des œstrogènes incorporant de nouveaux liants identifiés à l'aide d'un criblage de chimiothèques codées sur l'ADN. Journal of Medicinal Chemistry 2021, 64 (8), 5049-5066.


